특정 종의 Annotation 파일을 다운 받는 방법
Annotation 파일에는 각 유전자가 어떤 유전자인지에 대한 정보가 들어있다.
1. 아래의 Ensembl 홈페이지로 들어간다.
https://asia.ensembl.org/index.html
Ensembl genome browser 100
Tools All tools BioMart > Export custom datasets from Ensembl with this data-mining tool BLAST/BLAT > Search our genomes for your DNA or protein sequence Variant Effect Predictor > Analyse your own variants and predict the functional consequences of known and unknown variants Search ...
asia.ensembl.org
2. BioMart 클릭
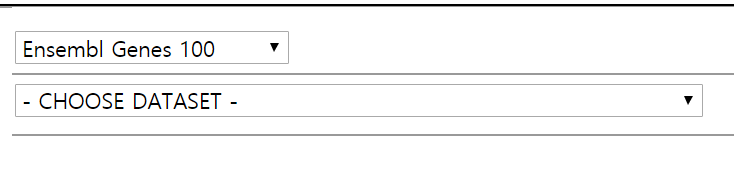
3. CHOOSE DATABASE 클릭 하여 Ensembl Genes 100 선택

4. CHOOSE DATASET 선택하여 원하는 종을 클릭한다.
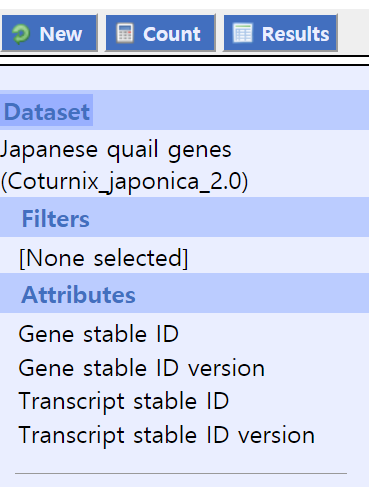
5. 왼쪽의 Attributes 메뉴 클릭
6. GENE: 부분에서 원하는 정보들을 클릭한 후,
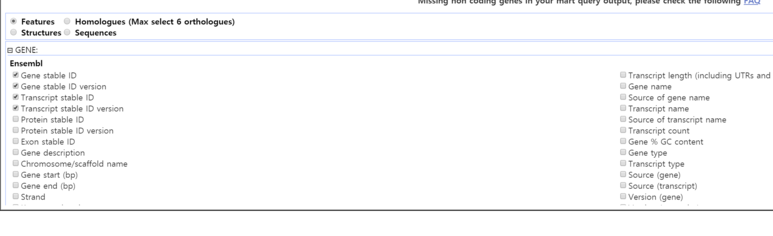
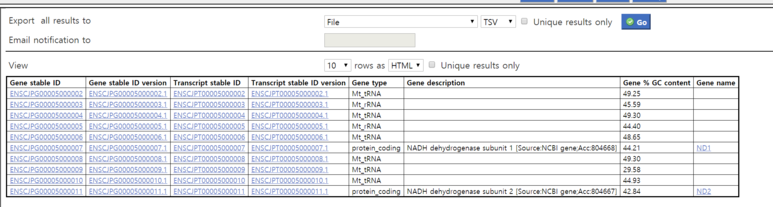
7, 왼쪽 위에 Results 버튼을 클릭한다. 그 후, 아래에 대략적인 표의 모습이 보인다.
그 후 Export all results to 파일 형식을 정하면 파일이 다운로드가 진행된다.
주의할 점
데이터베이스에서 가져오는 것이므로 버전이 바뀔 수 있으므로 다운받을 때마다 해당 유전자의 버전으로 이름을 저장해두는게 좋다.
'공부 > 바이오인포매틱스' 카테고리의 다른 글
| NGS란? (0) | 2022.09.05 |
|---|---|
| 시퀀싱이란? (0) | 2021.10.11 |
| [바이오인포매틱스] Fastq 파일형식 (0) | 2021.10.06 |
| [바이오인포매틱스] SRAtoolkit 설치 (0) | 2021.10.06 |
| [Sequence assembly] Overlap Consensus Graph (0) | 2021.09.13 |